当前位置:网站首页>论文解读:《i4mC-Deep: 利用具有化学特性的深度学习方法,对 N4-甲基胞嘧啶位点进行智能预测》
论文解读:《i4mC-Deep: 利用具有化学特性的深度学习方法,对 N4-甲基胞嘧啶位点进行智能预测》
2022-07-23 05:43:00 【风灬陌】
i4mC-Deep: An Intelligent Predictor of N4-Methylcytosine Sites Using a Deep Learning Approach with Chemical Properties
文章链接:https://www.mdpi.com/2073-4425/12/8/1117
DOI:https://doi.org/10.3390/genes12081117
期刊:Genes(三区)
影响因子:4.096
发布时间:2021年7月23日
服务器:http://nsclbio.jbnu.ac.kr/tools/i4mC-Deep/
补充文件: https://www.mdpi.com/article/10.3390/genes12081117/s1
代码和数据:https://github.com/waleed551/i4mC-Deep
1.文章概述
DNA受到N4-甲基胞嘧啶(4mC)分子的表观遗传修饰。N4-甲基胞嘧啶在DNA修复和复制中起重要作用,保护宿主DNA免受降解,调节DNA表达。目前的实验技术昂贵又费力。传统的基于机器学习的方法依赖于手工提取的特征,但是新方法通过利用学习特性而节省了时间和计算成本。在这项研究中,我们提出了i4mC-Deep,这是一个基于卷积神经网络(CNN)的智能预测器,可以预测DNA样本中的4mC修饰位点。提取DNN序列的核苷酸化学特性和核苷酸密度特征,作为CNN的输入数据。提出的方法的结果优于几个最先进的预测器。用i4mC-Deep方法分析地下黑麦草DNA,与传统预测相比,准确率(ACC)提高了3.9%,MCC提高了10.5% 。
2.背景
最近,一些计算工具已经被开发用于识别4mC位点,包括iDNA4mC,4mCPred,4mCPred-SVM和SOMM4mC。所有这些工具都是基于机器学习技术和手工制作的功能。iDNA4mC使用核苷酸化学特性和核苷酸频率作为特征向量结合支持向量机(SVM)来检测4mC位点。4mCPred和4mCPred-SVM也使用的是支持向量机,但有不同的特征表示,4mCPred利用两种特征编码技术,即位置特异性三核苷酸倾向(PSTNP)和三核苷酸的电子-离子相互作用;4mCPred-SVM将四种特征应用于4mC位点的组合预测,即K-mer 二核苷酸频率、单核苷酸二进制编码、二核苷酸二进制编码和局部位置特异性二核苷酸频率。SOMM4mC应用经典的一阶和二阶马尔可夫模型来预测4mC表观遗传修饰位点,并显示出比前面提到的其他工具更好的性能。此外,4mCCNN和DeepTorrent是基于深度学习的技术。4mCCNN采用One-hot编码的数据表示和卷积神经网络。DeepTorrent使用了四种带卷积和LSTM层的特征提取技术。以往的深度学习模型采用复杂结构,增加了参数和计算量。
在这项研究中,作者使用了一个卷积神经网络(CNN)来开发一个精确而有效的计算工具。CNN包括:卷积层(convolution)、批次归一化层(batch normalization)、扁平化层(Flatten)、丢失层(Dropout)和全连接层(Dense),卷积层用于自动提取编码的DNA序列中的重要特征。作者用核苷酸化学性质(NCP)和核苷酸密度(ND)方法编码输入的DNA序列,然后使用批次归一化和Dropout控制过拟合,最后利用全连接层将DNA序列分为4mC位点和非4mC位点。使用10倍交叉验证技术来评估 i4mC-Deep,i4mC-Deep的结果优于以前的工具。i4mC-Deep的架构如图1所示。作者还开发了一个免费的在线网络服务器。
2.数据
数据集在开发高效可靠的计算工具方面起着非常重要的作用。作者利用了6个不同种类的原核生物和真核生物、Caenorhabditis elegans, Drosophila melanogaster, Arabidopsis thaliana, Escherichia coli, Geoalkalibacter subterraneus, and Geobacter pickeringii.的数据。这些数据集是使用MethSMRT数据库构建的。基准数据集包括1554、1769、1978、388、906和569个阳性和阴性样本。六个数据集中的每个序列都有一个位于中心的胞嘧啶 ,长度为41碱基。
3.方法
3.1 特征编码
- 核苷酸化学性质 (nucleotide chemical properties,NCP)

- 核苷酸密度(nucleotide density,ND)
DNA序列中每个核苷酸的频率信息。
3.2 模型
参数选择范围:
最佳参数:卷积层为2,两层滤波器大小为8,两层的填充量为“same”,两层的内核大小为3,丢失概率为0.3。
应用 l2正则化和 dropout 正则化来避免网络的过拟合,使用学习率为0.001的Adam优化器,batch size最佳为32,设置的迭代次数(epochs)为200,可提前停止。
4.结果
4.1 与其他最先进方法比较
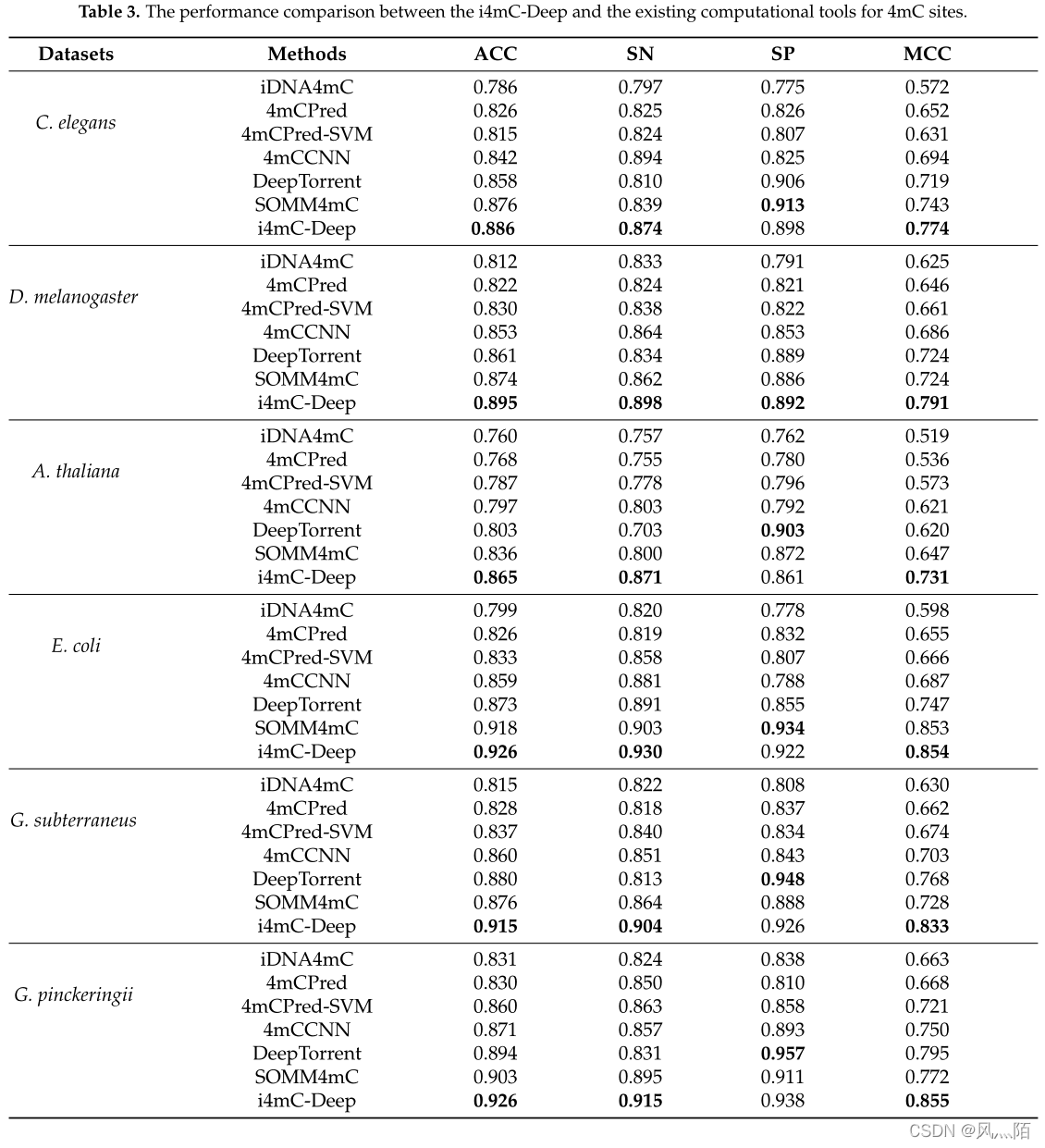

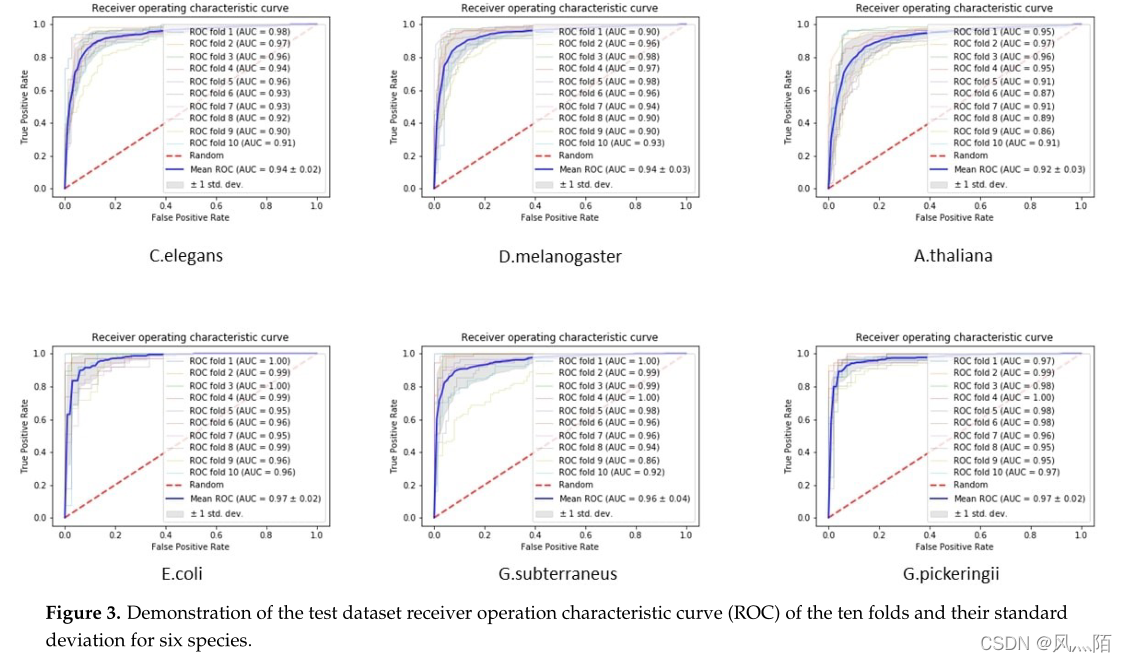
4.2 序列分析
t-SNE可视化: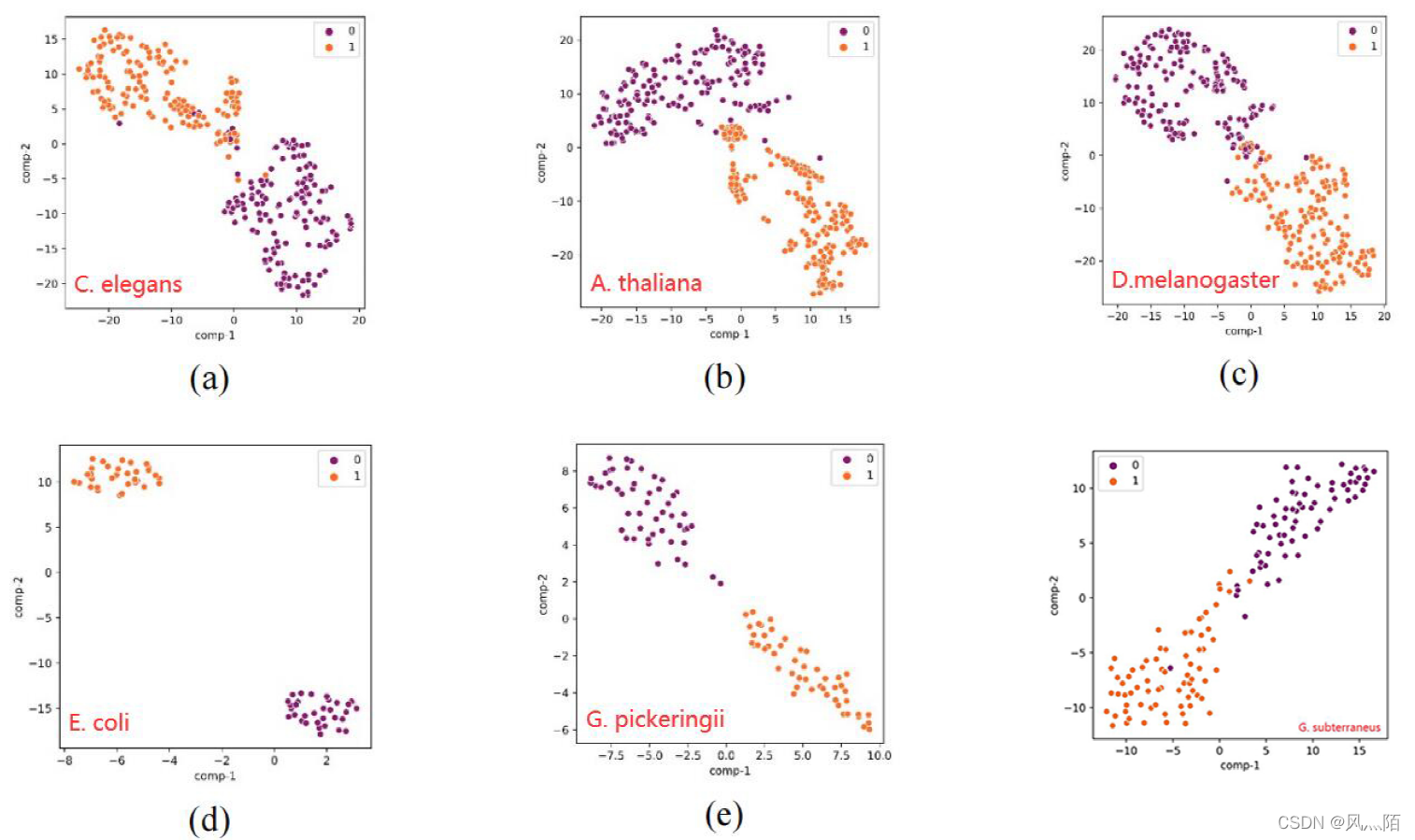
电子突变分析中的热图:

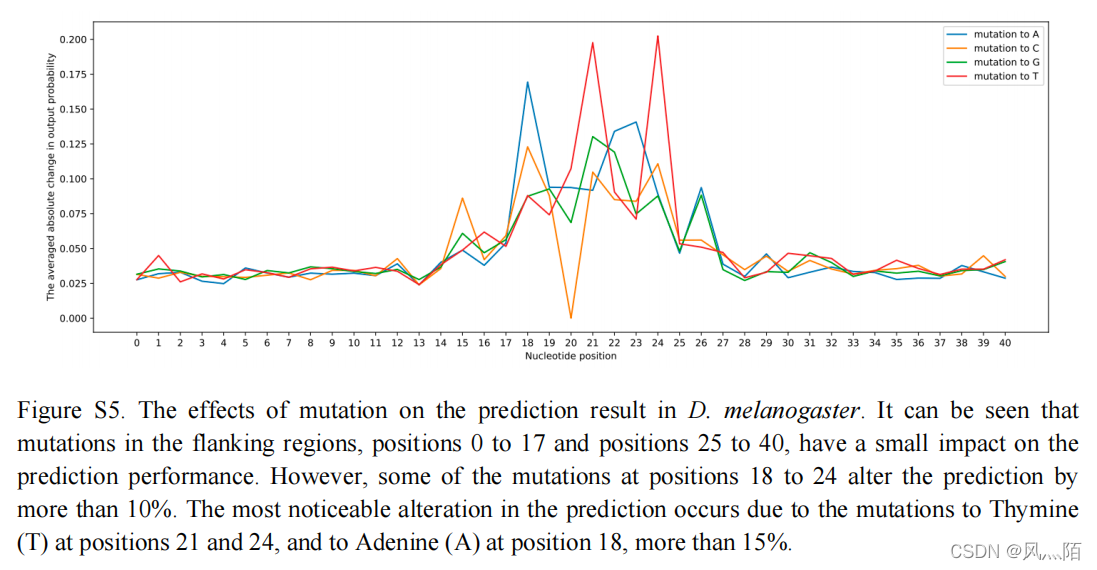
突变对预测概率的影响: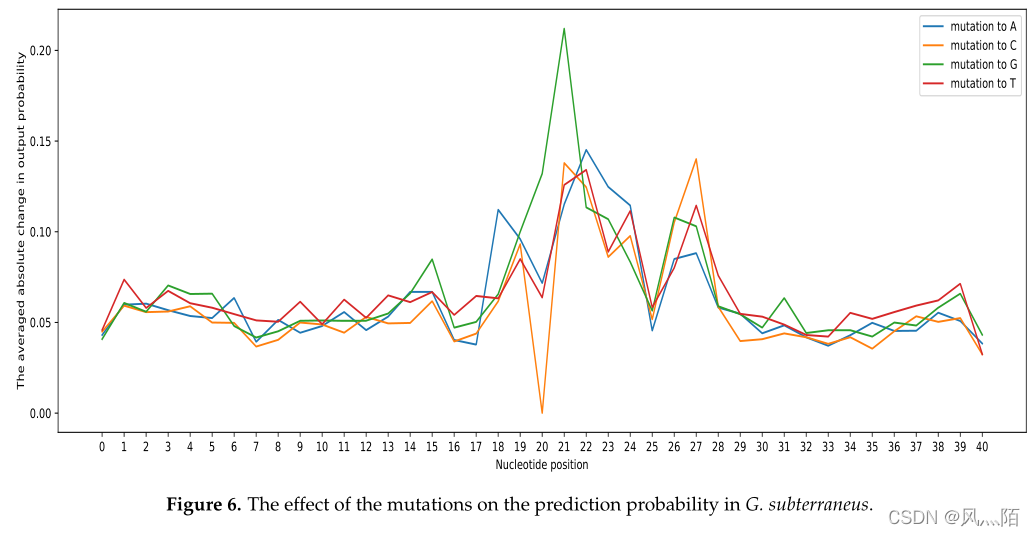




5.Web服务器
链接:http://nsclbio.jbnu.ac.kr/tools/i4mC-Deep/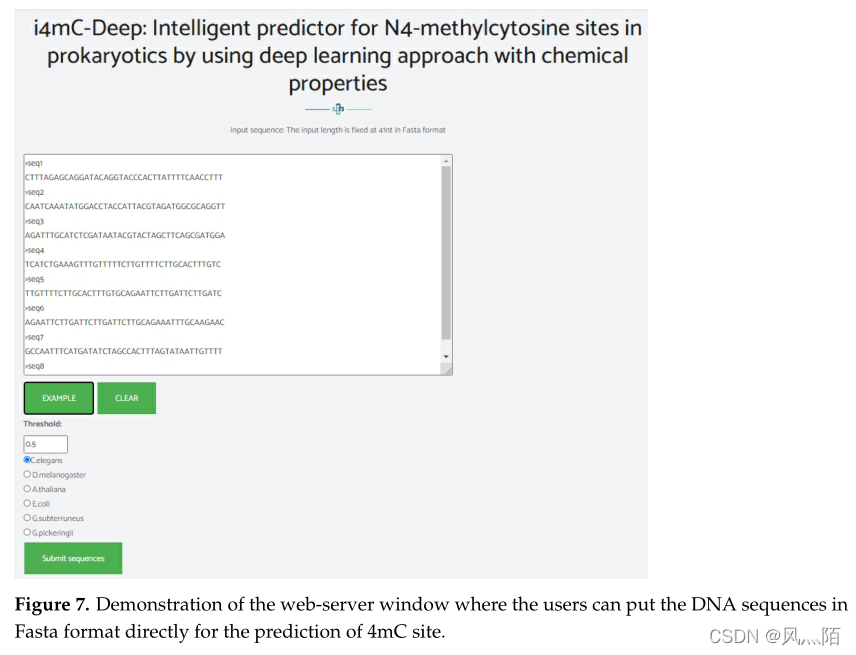

边栏推荐
猜你喜欢
Upload pictures to qiniu cloud through the web call interface

对.h5文件的迭代显示,h5py数据操作

Gartner调查研究:中国的数字化发展较之世界水平如何?高性能计算能否占据主导地位?

Vio --- boundary adjustment solution process

2、MySQL数据管理--DML(添加、修改、删除数据)

NVIDIA 英伟达发布H100 GPU,水冷服务器适配在路上

知识图谱、图数据平台、图技术如何助力零售业飞速发展
查看真机APP里面沙盒文件

飞桨高层API实现图像去雨

New BPMN file used by activiti workflow
随机推荐
实用卷积相关trick
Standardize database design
MySQL卸载
Circular queue
对.h5文件的迭代显示,h5py数据操作
Mysql database
可能逃不了课了!如何使用paddleX来点人头?
UE4解决WebBrowser无法播放H.264的问题
方法的定义应用
从已有VOC2007数据集生成yolov3所需要的数据集,以及正式开始调试程序需要修改的地方
Accordion effect
NVIDIA 英伟达发布H100 GPU,水冷服务器适配在路上
with语句
Gerrit operation manual
Vio --- boundary adjustment solution process
高德定位---权限弹框不出现的问题
UE4 solves the problem that the WebBrowser cannot play H.264
Maybe I can't escape class! How to use paddlex to point the head?
Hard disk partition of obsessive-compulsive disorder
Static linked list